给出一个长度为10的由基因“ATGC”随机组成的参照字符串,然后再给出一个长度为6的可操作基因字符串,可操作字符串的字符顺序不能变,但是可以移动其位置,求移动字符串和参照字符串的最大配对率
题目来源
项目中的一个”基因“配对小游戏,如下: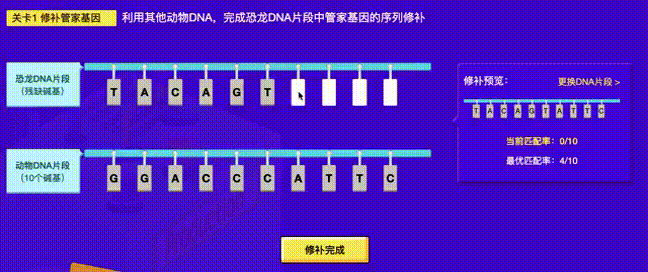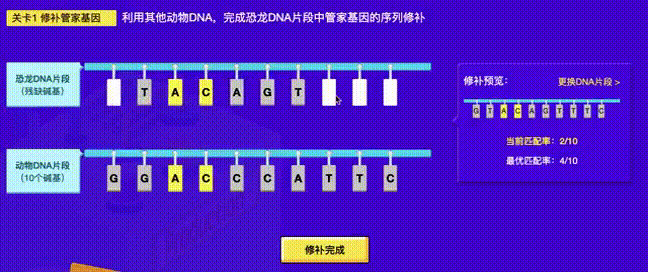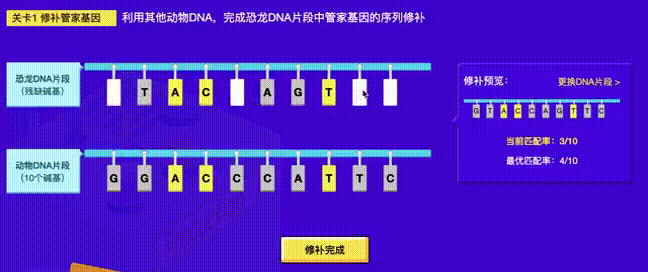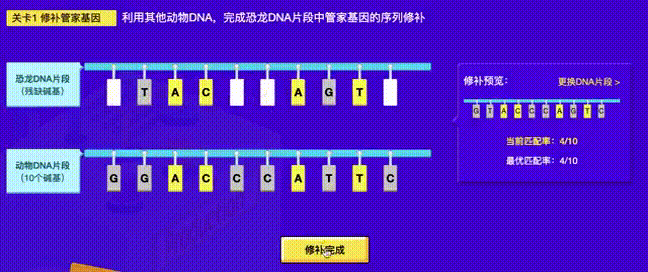
按上图中给出的例子:
- 参考碱基序列为:
GGACCCATTC - 可顺序移动碱基序列为:
TACAGT**** - 其中
*可移动,匹配率 = 符合配对规则的字母个数 / 总字母个数 - 当可移动碱基的位置为
*TAC**AGT*时有和参考序列有4个碱基匹配,此时匹配率为:4/10,是最优匹配率
全组合
为了节省时间,项目中采取了全组合的方法,把所有情况列举出来,然后对比,取匹配率最高的一种情况作为答案。 转换一下就知道,题目等于是有10个位置,任意取四个放空白碱基,一共有C(10,4) = 10!/(4! * 6!) = 10 * 9 * 8 * 7 / (1 * 2 * 3 * 4) = 210种情况,代码如下:
1 | function toCalculateBestPairingRatio() { |
动态规划
全组合虽然能够解决本次问题,但是其复杂度为Ο(n!),当基数再大一点的时候就不适合了。像这种求解最优的问题非常适合使用动态规划,把上题中的基因通过表格列出,行表示参考碱基序列,列表示可顺序移动碱基序列,从第一个碱基开始逐渐增加,表格内为最佳碱基配对率
| j\i | G | G | A | C | C | C | A | T | T | C |
|---|---|---|---|---|---|---|---|---|---|---|
| T | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 |
| A | - | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| C | - | - | 0 | 2 | 2 | 2 | 2 | 2 | 2 | 2 |
| A | - | - | - | 0 | 2 | 2 | 3 | 3 | 3 | 3 |
| G | - | - | - | - | 0 | 2 | 2 | 3 | 3 | 3 |
| T | - | - | - | - | - | 0 | 2 | 3 | 4 | 4 |
Row表示行,i为其下标,Col表示列,j为其下标,Rate表示最佳匹配率,通过观察可以发现规律:
Rate[j][i] = Max{Rate[j-1][i-1] + Number(Row[i] == Col[j]), Rate[j][i - 1]}
其中当只有一行时Rate[j-1][i-1] = 0,表格左下角则为最大的匹配率
1 | function toCalculateBestPairingRatio() { |
该方法的算法复杂度为平方阶,能够完美解决当前问题,就算后面的基数扩增也不怕了